上海金畔生物科技有限公司代理AAT Bioquest荧光染料全线产品,欢迎访问AAT Bioquest荧光染料官网了解更多信息。
Helixyte Green双链DNA定量检测试剂盒
 |
货号 | 17646 | 存储条件 | Multiple |
| 规格 | 1000 Tests | 价格 | 6564 | |
| Ex (nm) | 503 | Em (nm) | 527 | |
| 分子量 | 溶剂 | |||
| 产品详细介绍 | ||||
简要概述
DNA定量是DNA样品制备过程中的一项重要工作,Helixyte 绿色双链DNA定量检测试剂盒提供了一种用Helixyte Green BR快速测定dsDNA的方法。该测定法在三个数量级上是线性的,并且比紫外线吸收度读数灵敏度高几个数量级。Helixyte Green-BR与dsDNA结合后荧光增强,序列依赖性小,可准确测定基因组DNA、病毒DNA、miniprep-DNA或PCR扩增产物等多种来源的DNA样品。该方法对RNA上的双链DNA(dsDNA)具有高度的选择性,并且被优化以测量从10 pg/ul到10 ng/ul的DNA浓度。金畔生物是AAT Bioquest的中国代理商,为您提供最优质的Helixyte 绿色双链DNA定量检测试剂盒。
点击查看光谱
适用仪器
| 荧光酶标仪 | |
| 激发: | 490nm |
| 发射: | 530nm |
| cutoff: | 515nm |
| 推荐孔板: | 黑色孔板 |
产品说明书
样品实验方案
简要概述
- 准备dSDNA标准品,测试样品和染料工作溶液
- 添加DNA标准液或测试样品(50 uL)
- 添加Helixyte Green-BR工作溶液(50 uL)
- 在室温下孵育2分钟
- 检测Ex / Em = 490/530 nm的荧光强度
提示:操作前将所有组件加热到室温。暂时没有关于Helixyte Green dsDNA染色剂的致突变性或毒性的数据。由于该试剂与核酸结合,因此应将其视为潜在的诱变剂,应谨慎处理。DMSO储备溶液应特别小心,因为已知DMSO有助于有机分子进入组织。
溶液配制
标准溶液配制
将100 uL的100 ug / mL dsDNA标准溶液(组分C)添加到400 uL的测定缓冲液(组分B)中,以生成20 ug / mL的dsDNA标准溶液。然后用测定缓冲液(组分B)进行1:3的系列稀释,以得到0至20 ug / mL的系列稀释的dsDNA标准品。
工作溶液配制
Helixyte Green BR工作溶液:将50 uL Helixyte Green BR(组分A)添加到5 mL的测定缓冲液(组分B)中,使总体积为5.050 mL。通过用箔纸覆盖或将其置于黑暗中来保护工作溶液免受光照。 注意:我们建议在塑料容器中而不是玻璃中制备该溶液,因为染料可能会吸附到玻璃表面。为了获得最佳结果,应在准备后的几个小时内使用此溶液。
实验步骤
表1. 在透明底部96孔微孔板中的dsDNA标准品和测试样品的布局。DS = dsDNA标准品(DS1-DS7,20至0.027 ug / mL); BL =空白对照;TS =测试样品
|
BL |
BL |
TS |
TS |
|
DS1 |
DS1 |
… |
… |
|
DS2 |
DS2 |
… |
… |
|
DS3 |
DS3 |
|
|
|
DS4 |
DS4 |
|
|
|
DS5 |
DS5 |
|
|
|
DS6 |
DS6 |
|
|
|
DS7 |
DS7 |
|
|
表2. 每个孔的试剂组成
|
Well |
Volume |
Reagent |
|
DS1-DS7 |
50 uL |
连续稀释 (20 to 0.027 ug/mL) |
|
BL |
50 uL |
缓冲液 (Component B) |
|
TS |
50 uL |
实验样品 |
- 根据表1和表2提供的布局,制备dsDNA标准品(DS)、空白对照品(BL)和测试样品(TS)。对于384孔板,每孔使用25 uL试剂,而不是50 uL。注:根据需要处理细胞或组织样本。
- 添加50 uL Helixyte Green-BR染料工作液(2X)分别加入dsDNA标准液、空白对照液和供试品中,使总分析体积为100ul/孔。对于384孔板,添加25 uL的Helixyte Green-BR染料工作溶液,每孔总体积为50 uL/孔。
- 在室温下避光培养2分钟。
- 在Ex/Em=490/530 nm(截止=515nm)处用荧光酶标仪检测荧光强度。
图示
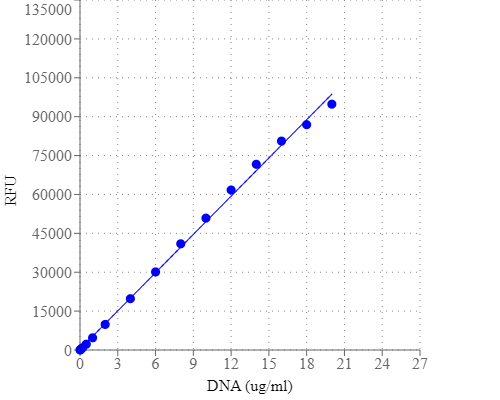
 图1.用Helixyte Green dsDNA定量试剂盒在96孔黑色孔板中测量了dsDNA剂量反应。 |
参考文献
A new reporter design based on DNA origami nanostructures for quantification of short oligonucleotides using microbeads
Authors: Choi, Y., Schmidt, C., Tinnefeld, P., Bald, I., Rodiger, S.
Journal: Sci Rep (2019): 4769
A universal fluorescence-based toolkit for real-time quantification of DNA and RNA nuclease activity
Authors: Sheppard, E. C., Rogers, S., Harmer, N. J., Chahwan, R.
Journal: Sci Rep (2019): 8853
Effects of Quantification Methods, Isolation Kits, Plasma Biobanking, and Hemolysis on Cell-Free DNA Analysis in Plasma
Authors: Streleckiene, G., Forster, M., Inciuraite, R., Lukosevicius, R., Skieceviciene, J.
Journal: Biopreserv Biobank (2019): ersion=”1.0″ encoding=”UTF-8″ ?>17645.enlEndNote1117Streleckiene, G.Forster, M.Inciuraite, R.Lukosevicius, R.Skieceviciene, J.1Institute for Digestive Research, Lithuanian University of Health Sciences, Kaunas, Lithuania. 2Institute of Clinical Molecu
Flat-top TIRF illumination boosts DNA-PAINT imaging and quantification
Authors: Stehr, F., Stein, J., Schueder, F., Schwille, P., Jungmann, R.
Journal: Nat Commun (2019): 1268
Molecular-Recognition-Based DNA Nanodevices for Enhancing the Direct Visualization and Quantification of Single Vesicles of Tumor Exosomes in Plasma Microsamples
Authors: He, D., Ho, S. L., Chan, H. N., Wang, H., Hai, L., He, X., Wang, K., Li, H. W.
Journal: Anal Chem (2019): 2768-2775
Quantification of fixed adherent cells using a strong enhancer of the fluorescence of DNA dyes
Authors: Ligasova, A., Koberna, K.
Journal: Sci Rep (2019): 8701
A fluorescent reporter for quantification and enrichment of DNA editing by APOBEC-Cas9 or cleavage by Cas9 in living cells
Authors: St Martin, A., Salamango, D., Serebrenik, A., Shaban, N., Brown, W. L., Donati, F., Munagala, U., Conticello, S. G., Harris, R. S.
Journal: Nucleic Acids Res (2018): e84
Accuracy of human sperm DNA oxidation quantification and threshold determination using an 8-OHdG immuno-detection assay
Authors: Vorilhon, S., Brugnon, F., Kocer, A., Dollet, S., Bourgne, C., Berger, M., Janny, L., Pereira, B., Aitken, R. J., Moazamian, A., Gharagozloo, P., Drevet, J., Pons-Rejraji, H.
Journal: Hum Reprod (2018): 553-562
Cell Type-Specific Quantification of Telomere Length and DNA Double-strand Breaks in Individual Lung Cells by Fluorescence In Situ Hybridization and Fluorescent Immunohistochemistry
Authors: van Batenburg, A. A., Kazemier, K. M., Peeters, T., van Oosterhout, M. F. M., van der Vis, J. J., Grutters, J. C., Goldschmeding, R., van Moorsel, C. H. M.
Journal: J Histochem Cytochem (2018): 485-495
Identification and Quantification of Heterogeneously-methylated DNA Fragments Using Epiallele-sensitive Droplet Digital Polymerase Chain Reaction (EAST-ddPCR)
Authors: Menschikowski, M., J and eck, C., Friedemann, M., Richter, S., Thiem, D., Lange, B. S., Suttorp, M.
Journal: Cancer Genomics Proteomics (2018): 299-312
